Investigadores de la Universidad de Stanford informan que el uso de ciprofloxacino genera una resistencia persistente a los antibióticos en las bacterias intestinales humanas, y que la resistencia surge de forma independiente en diversas especies y perdura durante más de 10 semanas.
por Justin Jackson, Medical Xpress
La resistencia a los antimicrobianos (RAM) es un problema de salud mundial vinculado a millones de muertes cada año. Se debe, en gran medida, al uso excesivo e inadecuado de antibióticos. Los estudios previos sobre la RAM se han basado principalmente en experimentos in vitro y modelos animales, que no logran reproducir la complejidad completa de los entornos microbianos humanos.
En el estudio titulado «El uso breve de antibióticos impulsa las bacterias intestinales humanas hacia una resistencia de bajo costo», publicado en Nature , los investigadores realizaron un estudio metagenómico longitudinal para explicar cómo evoluciona la resistencia in vivo.
Sesenta adultos sanos recibieron ciprofloxacino, 500 mg dos veces al día, durante cinco días. Durante un período de 20 semanas, los participantes recolectaron 16 muestras de heces, obteniendo 960 muestras para análisis. Se realizó una secuenciación metagenómica shotgun en todas las muestras, generando un promedio de 18,8 millones de lecturas por muestra. Se desarrolló una herramienta computacional llamada PolyPanner para identificar sitios polimórficos reales a lo largo del tiempo.
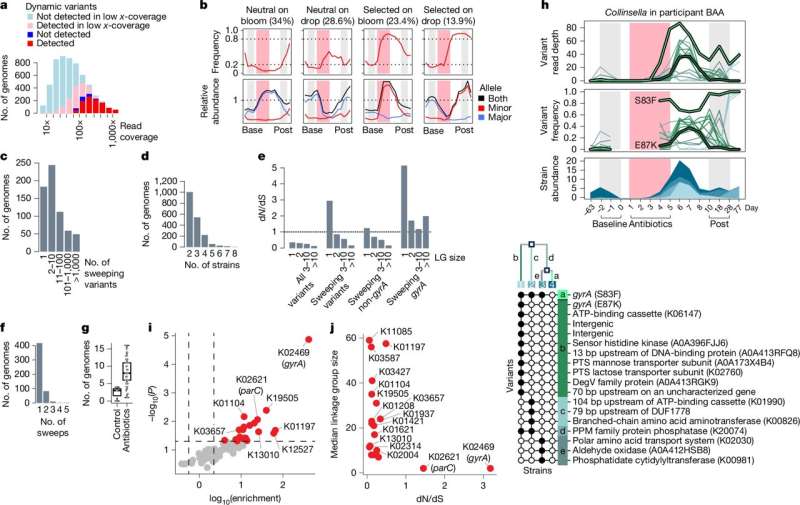
Los investigadores reconstruyeron 5665 genomas que representan poblaciones bacterianas comensales e identificaron 2,3 millones de variantes genéticas. Entre estas, 513 poblaciones mostraron barridos selectivos, una clara evidencia de evolución adaptativa. Se observó una alta concentración de mutaciones en gyrA, un gen asociado con la resistencia a las fluoroquinolonas.
Entre las 513 poblaciones en evolución, se produjeron con frecuencia cambios genéticos radicales en el gen gyrA, fundamental para la resistencia a las fluoroquinolonas. Sesenta y tres poblaciones de 34 participantes presentaron mutaciones en el gen gyrA, que generalmente surgieron de forma independiente en los individuos. Casi el 10 % de las poblaciones bacterianas inicialmente susceptibles adquirieron resistencia a través de estas mutaciones.
Una vez establecidos, los barridos de gyrA persistieron más de 10 semanas y se predijo que permanecerían detectables hasta un año. Se observaron mutaciones adicionales asociadas a la resistencia en otros genes, aunque estos eventos fueron menos frecuentes y se presentaron en menos especies.
La resistencia tuvo mayor probabilidad de surgir en poblaciones que eran abundantes antes del tratamiento y experimentaron disminuciones significativas durante la exposición, lo que identifica una condición correlacionada con mayores probabilidades de cambio evolutivo.
Las mutaciones de resistencia no conllevaron costos de adaptación, lo que permitió que las cepas resistentes mantuvieran una población dominante tras la finalización del tratamiento. La secuenciación dirigida no mostró evidencia de reversión de la resistencia. Las mutaciones en gyrA explicaron solo una parte de la resistencia observada, lo que sugiere mecanismos adicionales.
Según los resultados, incluso el uso de antibióticos a corto plazo puede provocar mutaciones de resistencia que persisten en el intestino humano durante meses tras la finalización del tratamiento. Las mutaciones surgen de forma independiente en las distintas especies bacterianas y no tienen un coste de adaptación medible, lo que permite que las cepas resistentes se mantengan prevalentes.
Se ha demostrado que los microbios intestinales pueden desarrollar resistencia sin infección previa. Por lo tanto, las poblaciones comensales podrían actuar como reservorios de rasgos de resistencia que podrían transferirse a bacterias patógenas mediante transferencia horizontal de genes, más allá de la interacción con antibióticos.
Dado que la resistencia evolucionó de forma predecible según el tamaño de la población, es posible predecir los resultados de la resistencia si se conoce la población inicial antes del tratamiento. Se necesitan experimentos con diferentes combinaciones de poblaciones iniciales y tipos de tratamiento para ampliar completamente este modelo predictivo.
El monitoreo de la composición y abundancia microbiana antes y durante el tratamiento podría ayudar a orientar un uso más preciso de los antibióticos, reducir los riesgos a largo plazo asociados con la resistencia y mejorar la gestión general del uso de antibióticos.
Más información: Eitan Yaffe et al., El uso breve de antibióticos impulsa la resistencia de bajo coste en las bacterias intestinales humanas, Nature (2025). DOI: 10.1038/s41586-025-08781-x
Leyenda foto principal: Variantes, cepas, LG y análisis de evolución convergente. Crédito: Nature (2025). DOI: 10.1038/s41586-025-08781-x
Nota editorial:
Este artículo ha sido elaborado con fines divulgativos a partir de información pública y fuentes especializadas, adaptado al enfoque editorial del medio para facilitar su comprensión y contextualización.
